Substrate Specificity in Promiscuous Enzymes: The Case of the Aminoaldehyde Dehydrogenases from Pseudomonas aeruginosa
DOI:
https://doi.org/10.29356/jmcs.v67i3.2022Keywords:
Aldehyde dehydrogenase, physiological substrate, structure-function relationships, active-site residues, active-site conformationAbstract
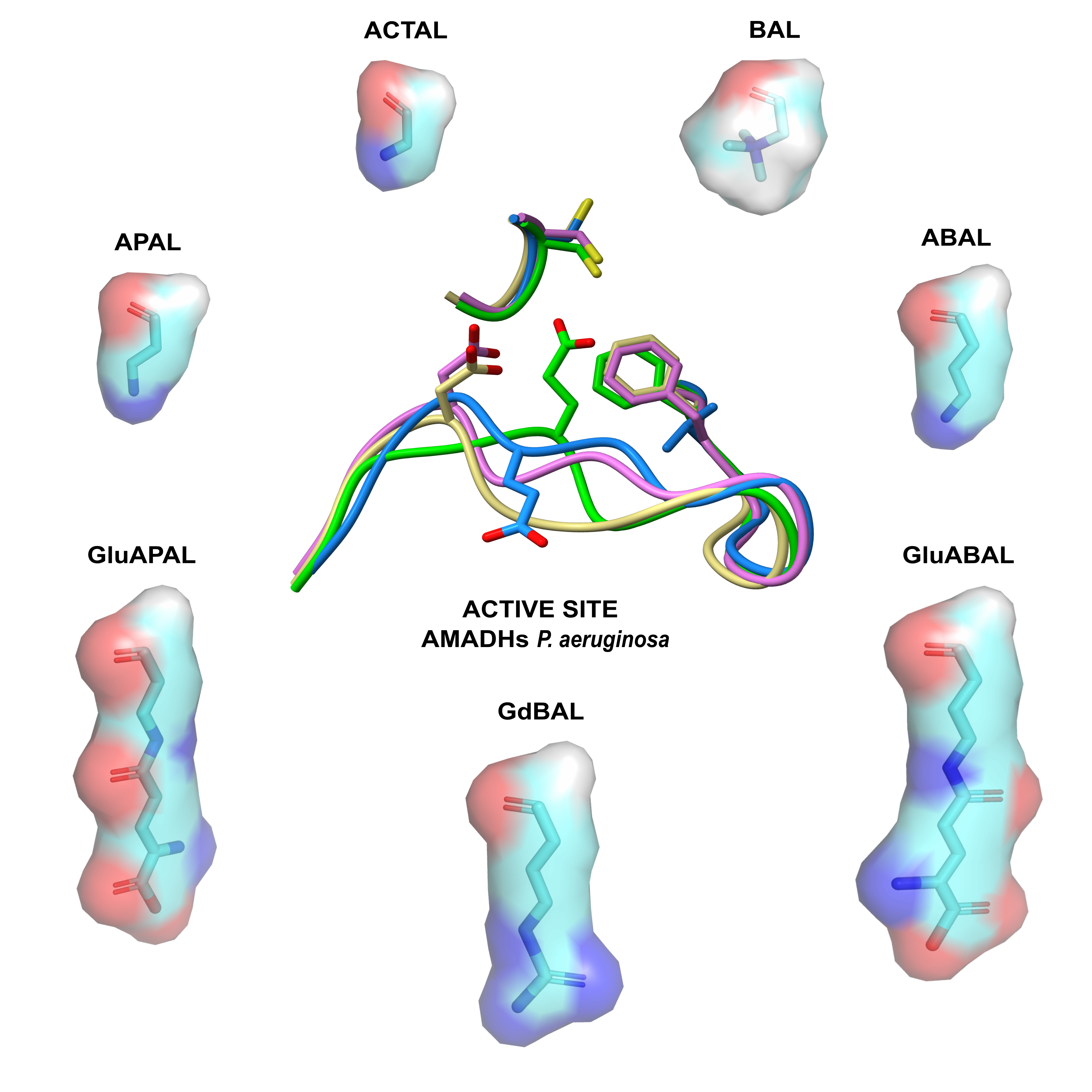
Abstract. Substrate specificity is instrumental in enzyme catalysis and a major determinant of the enzyme’s physiological role. Nevertheless, the degree of substrate specificity may vary in an ample range; some enzymes exhibit very high specificity while others show a relaxed specificity or promiscuity. The latter is used by evolution for the emergence of new enzymes able to perform novel metabolic roles. The basis of substrate specificity is substrate recognition, which is achieved in the enzyme active site by chemical and structural mechanisms. Here, we exemplify that specificity may exist within promiscuity by comparatively analyzing kinetic and structural data of the four Pseudomonas aeruginosa aminoaldehyde dehydrogenases—PA5373, PA5312, PA4189, and PA0219. Despite their apparent substrate promiscuity, we found that these enzymes show a significant degree of substrate specificity. They have evolved to preferentially oxidize different aminoaldehydes, even though each of them can use as in vitro substrates most of the aminoaldehydes preferred by the others. We focus on the role played in these enzymes by two active-site residues—one acidic and the other aromatic, both belonging to the so-called “anchor” loop—in binding the aldehyde amino group, as well as on the importance of the anchor loop conformation in defining the size and shape of the active-site cavity. Our results support the notion that natural selection has fine-tuned the active-site structural and chemical features of the P. aeruginosa AMADH enzymes to the structural and chemical features of their physiological aminoaldehydes substrates.
Resumen. La especificidad de sustrato es fundamental para la catálisis enzimática y un importante determinante del papel fisiológico de una enzima. Sin embargo, el grado de especificidad de las enzimas puede variar en un amplio intervalo; algunas enzimas exhiben una especificidad estricta por sus sustratos mientras que otras exhiben una especicificidad relajada o promiscuidad. Esto último es utilizado por la evolución para que emerjan nuevas enzimas capaces de llevar a cabo funciones metabólicas novedosas. La base de la especificidad es el reconocimiento del sustrato, lo que se logra en el sitio activo de la enzima mediante mecanismos químicos y estructurales. En este trabajo mostramos que la especificidad puede existir dentro de la promiscuidad, analizando resultados cinéticos y estructurales de las cuatro aminoaldehído deshidrogenasas de Pseudomonas aeruginosa—PA5373, PA5312, PA4189 y PA0219. A pesar de su aparente promiscuidad, encontramos que estas enzimas presentan un alto grado de especificidad. Han evolucionado para oxidar preferencialmente algunos aminoaldehídos, aunque cada una de ellas pueda in vitro usar como sustratos la mayoría de aminoaldehídos preferidos por las otras. Enfocamos nuestro análisis en el papel que desempeñan dos residuos del sitio activo—uno ácido y el otro aromático, ambos pertenecientes a la llamada asa “ancla”—en la unión de los grupos aminos de sus aldehídos sustrato, así como en la importancia de la conformación de esta asa para definir el tamaño y la forma de la cavidad del sitio activo. Nuestros resultados apoyan la idea de que la selección natural ha finamente ajustado las características químicas y estructurales del sito activo de las AMADHs a las características químicas y estructurales de sus sustratos aldehídos fisiológicos.
Downloads
References
De Luca, V; Mandrich, L. Protein Pept. Lett. 2020, 27, 400-410. DOI: http://dx.doi.org/10.1021/ja055137+.
Vasiliou, V.; Pappa, A.; Petersen, D. R. Chem.-Biol. Interact. 2000, 129, 1–19. DOI: https://doi.org/10.1016/s0009-2797(00)00211-8.
Julián-Sánchez, A.; Riveros-Rosas, H.; Martínez-Castilla, L. P.; Velasco-García, R.; Muñoz-Clares, R. A. in: Enzymology and Molecular Biology of Carbonyl 13, H. Weiner, E. Maser, R. Lindahl, B. Plapp Ed., University Press, West Lafayette, IN, 2007, 64–76.
Riveros-Rosas, H.; Julián-Sánchez, A.; Moreno-Hagelsieb, G.; Muñoz-Clares, R. A. Chem.-Biol. Interact. 2019, 304, 83–87. DOI: https://doi.org/10.1016/j.cbi.2019.03.00.
Riveros-Rosas, H.; González-Segura, L.; Julián-Sánchez, A.; Díaz-Sánchez, A. G.; Muñoz-Clares, R. A. Chem.-Biol. Interact. 2013, 202, 51–61. DOI: https://doi.org/10.1016/j.cbi.2012.11.
Velasco-García, R.; Mújica-Jiménez, C.; Mendoza-Hernández, G.; Muñoz-Clares, R. A. J. Bacteriol. 1999, 181, 1292–1300. DOI: https://doi.org/10.1128/JB.181.4.1292-1300.1999.
Sage, A. E.; Vasil, A. I.; Vasil, M. L. Mol. Microbiol. 1997, 23, 43–56 DOI: https://doi.org/10.1046/j.1365-2958.1997.1681542.x.
Velasco-García, R.; Villalobos, M. A.; Ramírez-Romero, M. A. Mújica-Jiménez, C; Iturriaga, G.; Muñoz-Clares, R. A. Arch. Microbiol. 2006, 185, 14–22. DOI: https://doi.org/10.1007/s00203-005-0054-8.
Lu, C. D.; Itoh, Y.; Nakada, Y.; Jiang, Y. J. Bacteriol. 2002, 184, 3765–3773. DOI: https://doi.org/10.1128/JB.184.14.3765-3773.2002.
Yao, X.; He, W.; Lu, C. D. J. Bacteriol. 2011, 193, 3923–3930. DOI: https://doi.org/10.1128/JB.05105-11.
Cardona-Cardona, Y. V.; Regla, I.; Juárez-Díaz, J. A.; Carrillo-Campos, J.; López-Ortiz, M.; Aguilera-Cruz, A.; Mújica-Jiménez, C.; Muñoz-Clares, R. A. FEBS J., 2022, 289, 2685–2705. DOI: https://doi.org/10.1111/febs.16277.
Jann, A.; Matsumoto, H.; Haas D. J. Gen. Microbiol. 1988, 134, 1043–1053. DOI: https://doi.org/10.1099/00221287-134-4-1043.
Johnson, D. A.; Tetu, S. G.; Phillippy, K.; Chen, J.; Ren, Q.; Paulsen, I. T. PLoS Genet. 2008, 4, 1–11. DOI: https://doi.org/10.1371/journal.pgen.1000211.
Wang, Y.; Cao, Q.; Cao, Q.; Gan, J.; Sun, N.; Yang, C. G.; Bae, T.; Wu, M.; Lan, L. Sci. Bull. 2021, 66, 1101–1118. DOI: https://doi.org/10.1016/j.scib.2021.01.002.
Fernández-Silva, A.; Juárez-Vázquez, A. L.; González-Segura, L.; Juárez-Díaz, J. A.; Muñoz-Clares, R. A. Biochem. J. 2023, 480, 259–281. DOI: https://doi.org/10.1042/BCJ20220567.
Patel, B.; Percivalle, C.; Ritson, D.; Duffy, C. D.; Sutherland, J. D. Nature Chem. 2015, 7, 301–307. DOI: https://doi.org/10.1038/nchem.2202.
Laemmli, U. K. Nature. 1970, 227, 680–685. DOI: https://doi.org/10.1038/227680a0.
Gasteiger, E.; Gattiker, A.; Hoogland, C.; Ivanyi, I.; Appel R. D., Bairoch, A. Nucleic Acids Res. 2003, 31, 3784–3788. DOI: https://doi.org/10.1093/nar/gkg563.
Pettersen, E. F.; Goddard, T. D.; Huang, C. C.; Meng, E.C.; Couch, G. S.; Croll, T. I.; Morris, J. H.; Ferrin, T. E. Protein Sci. 2021, 30, 70–82. DOI: https://doi.org/10.1002/pro.3943.
Jumper, J.; Evans, R.; Pritzel, A.; Green, T; Figurnov, M; Ronneberger, O; Tunyasuvunakool, K; Bates, R. et al. Nature. 2021, 596, 583–589. DOI: https://doi.org/10.1038/s41586-021-03819-2.
Zhou, Y.; Wang, L.; Yang, F.; Lin, X.; Zhang, S.; Zhao, Z. K. Appl. Environ. Microbiol. 2011, 77, 6133–6140. DOI: https://doi.org/10.1128/AEM.00630-11.
Baek, M.; Dimaio, F.; Anishchenko, I.; Dauparas, J.; Ovchinnikov, S., Lee, G. R.; Wang, J.; Cong, Q. et al. Science. 2021, 373, 871–876. DOI: https://doi.org/10.1126/science.abj8754.
Farres, J.; Wang, X.; Takahashi, K.; Cunningham, S. J.; Wang, T. T. Y.; Weiner, H. J. Biol. Chem. 1994, 269, 13854–13860. DOI: https://doi.org/10.1016/S0021-9258(17)36725-X.
González-Segura, L.; Rudiño-Piñera, E.; Muñoz-Clares, R. A.; Horjales, E. J. Mol. Biol. 2009, 385, 542–57. DOI: https://doi.org/10.1016/j.jmb.2008.10.082.
Muñoz-Clares, R. A.; Díaz-Sánchez A. G.; González-Segura, L; Montiel, C. Arch. Biochem. Biophys. 2010, 493,71-81. DOI: https://doi.org/10.1016/j.abb.2009.09.006.
Arce-Rodríguez, A.; Volke, D. C.; Bense, S.; Häussler, S.; Nikel, P. I. Microb. Biotechnol. 2019, 12, 799–813. DOI: https://doi.org/10.1111/1751-7915.13439.
Riveros-Rosas, H.; González-Segura, L.; Julián-Sánchez, A. G.; Díaz-Sánchez, Muñoz-Clares, R. A. Chem.-Biol. Interact. 2013, 202, 51–61. DOI: https://doi.org/10.1016/j.cbi.2012.11.015.
Díaz-Sánchez, A. G.; González-Segura, L.; Mújica-Jiménez, C.; Rudiño-Piñera, E.; Montiel, C.; Martínez-Castilla, L. P.; Muñoz-Clares, R. A. Plant Physiol. 2012, 158, 1570–1582. DOI: https://doi.org/10.1104/pp.112.194514.
Pemberton, T. A.; Tanner, J. J. Arch. Biochem. Biophys. 2013, 538, 34–40. DOI: https://doi.org/10.1016/j.abb.2013.07.024.
Srivastava, D; Singh, R. K.; Moxley, M. A.; Henzl, M. T.; Becker, D. F.; Tanner, J. J. J. Mol. Biol. 2012, 420, 176–189. DOI: https://doi.org/10.1016/j.jmb.2012.04.010.
Campbell, A. C.; Bogner, A, N.; Mao, Y.; Becker, D. F.; Tanner, J. J. Arch. Biochem. Biophys. 2021, 15, 698–108727. DOI: https://doi.org/10.1016/j.abb.2020.108727.
Downloads
Published
Issue
Section
License
Copyright (c) 2023 Rosario A. Muñoz-Clares, Arline Fernández-Silva, Carlos Mújica-Jiménez, Sebastian Martínez-Flores

This work is licensed under a Creative Commons Attribution-NonCommercial 4.0 International License.
Authors who publish with this journal agree to the following terms:
- Authors retain copyright and grant the journal right of first publication with the work simultaneously licensed under a Creative Commons Attribution License that allows others to share the work with an acknowledgement of the work's authorship and initial publication in this journal.
- Authors are able to enter into separate, additional contractual arrangements for the non-exclusive distribution of the journal's published version of the work (e.g., post it to an institutional repository or publish it in a book), with an acknowledgement of its initial publication in this journal.