Biodiversity Assessment in Tecoma Species Using Micro-morphometric, Phytochemical and Molecular Descriptors Aided by Multivariate Analysis
DOI:
https://doi.org/10.29356/jmcs.v66i4.1702Keywords:
Cluster analysis, micro-morphometric features, OPLS-DA, TecomaAbstract
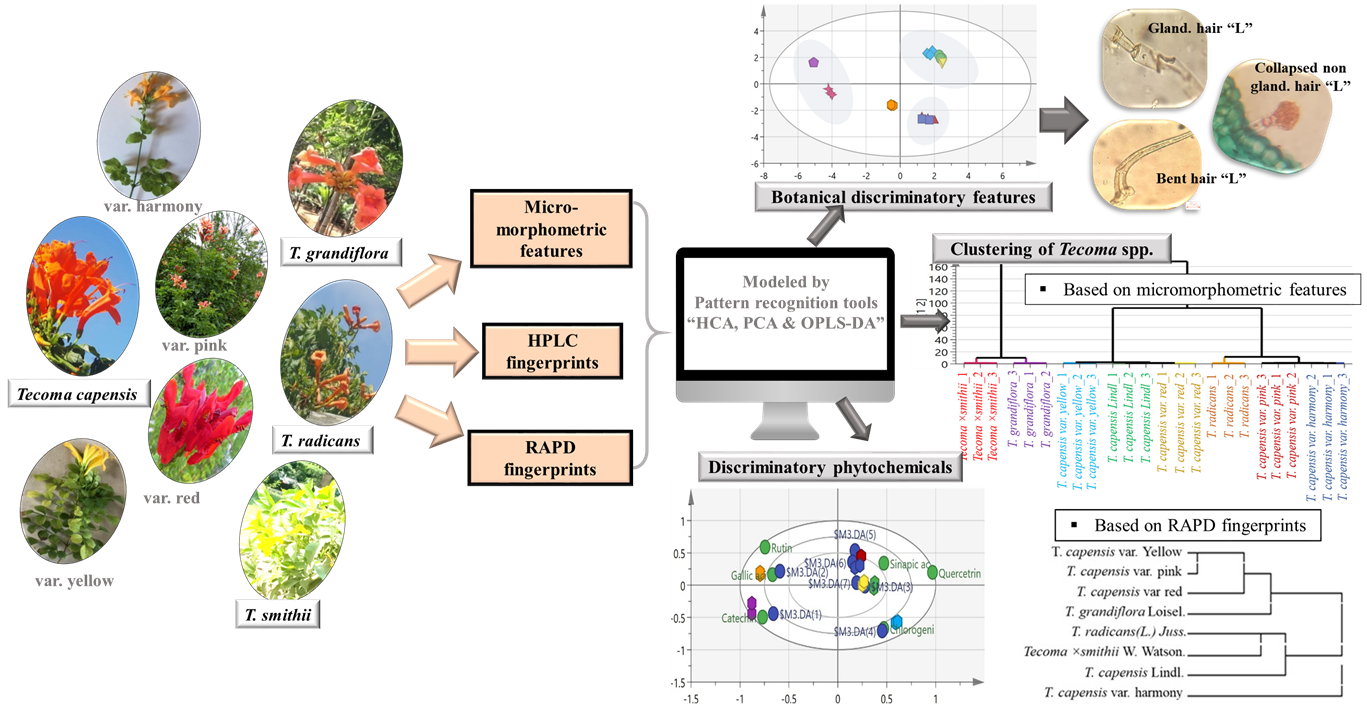
Abstract. Tecoma genus is a medicinally-valuable member in Bignoniaceae family comprising fourteen defectively-demarcated species with overlapping vegetative characters. Our study aimed to explore the micro-morphometric, phytochemical and genetic diversity among eight cultivated Tecoma plants. The variabilities existing in three data matrices derived from micro-morphometric features of powdered leaves, high performance liquid chromatographic fingerprints and Random Amplified Polymorphic DNA (RAPD) fingerprints were explored by multivariate analysis. The microscopical measurements of non-glandular trichomes, i.e. collapsed multicellular and branched, and glandular trichomes were revealed to be the significant botanical discriminatory features of the powdered plant specimens, however species-specific phytochemical markers were identified to be gallic acid, catechin and chlorogenic acid for T. radicans, T. grandiflora, T. capensis var. yellow, respectively. Sinapic acid was enriched in T capensis and its varieties i.e. pink, red and harmony. RAPD primers i.e. OP-B02, OP-C04 and OP-K2 served as molecular descriptors producing the highest polymorphism among the plants. The variabilities within two trumpet creeper plants “T. radicans and T. grandiflora” were unveiled via their micro-morphometric features as well as genetic fingerprints. Tecoma hybrid “Tecoma ×smithii” was more micro-morphometrically similar to the Chinese “T. grandiflora.” than T. capensis which is one of its parent plants, and proved to be more genetically related to it. This is the report for biodiversity in botanical, chemical and genetic aspects of Tecoma plants which are essential for the comprehensive authentication and differentiation among the studied plants assisted by multivariate analysis. The presented approach provides a competent and time-saving tool for simultaneously-discriminating among closely-related species.
Resumen. El género Tecoma es un miembro medicinalmente valioso de la familia Bignoniaceae que comprende catorce especies demarcadas defectuosamente con caracteres vegetativos superpuestos. Nuestro estudio tuvo como objetivo explorar la diversidad micromorfométrica, fitoquímica y genética entre ocho plantas de Tecoma cultivadas. Se exploraron mediante análisis multivariado las variabilidades existentes en tres matrices de datos derivadas de las características micromorfométricas de las hojas en polvo, las huellas dactilares por medio de cromatografía líquida de alta resolución y las huellas dactilares de ADN polimórfico amplificado al azar (RAPD). Las mediciones microscópicas de tricomas no glandulares, esto es, tricomas multicelulares colapsados y ramificados, y tricomas glandulares mostraron ser las características significativas de discriminación botánica de las muestras en polvo de plantas. Se identificaron marcadores fitoquímicos específicos para algunas especies como ácido gálico, catequina y ácido clorogénico para T. radicans, T. grandiflora, T. capensis var. amarillo, respectivamente. El ácido sinápico está enriqueció en T capensis y sus variedades, esto es, rosa, rojo y armonía. Los cebadores RAPD, esto es, OP-B02, OP-C04 y OP-K2 sirvieron como descriptores moleculares que producen el polimorfismo más elevado entre las plantas. A través de sus características micromorfométricas y sus huellas genéticas se determinó la variabilidad en dos plantas trepadoras de trompeta “T. radicans y T. grandiflora.” El híbrido de Tecoma “Tecoma × smithii” es micromorfométricamente más similar al chino “T. grandiflora” que a T. capensis, que es una de sus plantas progenitoras, y mostró estar más relacionada genéticamente con ella. En esta investigación de biodiversidad en aspectos botánicos, químicos y genéticos de las plantas de Tecoma que son esenciales para la autenticación y diferenciación integral entre las plantas estudiadas con la ayuda del análisis multivariado. El enfoque presentado proporciona una herramienta eficiente y que ahorra tiempo para discriminar simultáneamente entre especies estrechamente relacionadas.
Downloads
References
Wood, A. Trop. Plant Pathol. 2014, 39, 284–293. DOI: https://doi.org/10.1590/S1982-56762014000400003
Alarcon-Aguilara, F. J.; Roman-Ramos, R.; Perez-Gutierrez, S.; Aguilar-Contreras, A.; ContrerasWeber, C. C.; Flores-Saenz, J. L. J. Ethnopharmacol. 1998, 61, 101–110. DOI: https://doi.org/10.1016/S0378-8741(98)00020-8
Saini, N. K.; Singhal, M.; Srivastava, B. Chin. J. Nat. Med. 2012, 10, 138–141. DOI: https://doi.org/10.3724/SP.J.1009.2012.00138
Benzie, I. F. F.; Wachtel-Galor, S. NCBI Bookshelf. 2015, 1–11.
Contreras, R. N.; Ruter, J. M.; Conner, J.; Zeng, Y.; Ozias-Akins, P. Genome. 2012, 55, 437–445. DOI: https://doi.org/10.1139/g2012-030
Wood, J. R. I. A. Bot. J. Linn. Soc. 2008, 156, 143–172. DOI: https://doi.org/10.1111/j.1095-8339.2007.00731.x
Joselin, J.; Brintha, T. S. S.; Florence, A. R.; Jeeva, S. J. Chem. Pharm. Res. 2013, 5, 106–111.
Rosenshield, G.; Prokhorov, A. M. Slav. East Eur. J. 2006, 19, 366. DOI: https://doi.org/10.2307/306309
Verloove, F.; Salas-Pascual, M.; Rodríguez, Á. M. Flora Mediterr. 2018, 28, 119–135. DOI: https://doi.org/10.2985/026.025.0110
Kobayashi, N.; Hagiwara, J. C.; Miyajima, I.; Facciuto, G.; Soto, S.; Mata, D.; Escandon, A. J. Japanese Soc. Hortic. Sci. 2004, 73, 69–71. DOI: https://doi.org/10.2503/jjshs.73.69
Ganie, S. H.; Upadhyay, P.; Das, S.; Prasad Sharma, M. Plant Gene. 2015, 83–99. DOI: https://doi.org/10.1016/j.plgene.2015.10.002
Saleh, N. A.; Ezzat, S. M.; El-Kashoury, E. S. A.; Taha, K. F. J. Pharm. Sci. 2019, 44, 107–118.
Hegazi, N. M.; Khattab, A. R.; Frolov, A.; Wessjohann, L. A.; Farag, M. A. Food Chem. 2022, 367. DOI: https://doi.org/10.1016/j.foodchem.2021.130739
Farag, M. A.; Khattab, A. R.; Shamma, S.; Afifi, S. M. Foods. 2021, 10. DOI: https://doi.org/10.3390/foods10040728
Farag, M. A.; Afifi, S. M.; Rasheed, D. M.; Khattab, A. R. J. Food Compos. Anal. 2021, 102. DOI: https://doi.org/10.1016/j.jfca.2021.104073
Younis, I. Y.; Khattab, A. R.; Selim, N. M.; Sobeh, M.; Elhawary, S. S.; Bishbishy, M. H. E. Sci. Rep. 2022, 12. DOI: https://doi.org/10.1038/s41598-022-08479-4
Nour, V.; Trandafir, I.; Cosmulescu, S. Sci. 2013, 51, 883–890. DOI: https://doi.org/10.1093/chromsci/bms180
Williams, J. G. K.; Kubelik, A. R.; Livak, K. J.; Rafalski, J. A.; Tingey, S. V. Res. 1990, 18, 6531–6535. DOI: https://doi.org/10.1093/nar/18.22.6531
Ferrante, A.; Mariani, L. Horticulturae. 2018, 4, 21. DOI: https://doi.org/10.3390/horticulturae4030021

Downloads
Additional Files
Published
Issue
Section
License
Copyright (c) 2022 Seham S. El Hawary, Amira S. El Senousy, Hanan S. Marzouk, Mariam G. A. Alex, Amira R. Khattab

This work is licensed under a Creative Commons Attribution-NonCommercial 4.0 International License.
Authors who publish with this journal agree to the following terms:
- Authors retain copyright and grant the journal right of first publication with the work simultaneously licensed under a Creative Commons Attribution License that allows others to share the work with an acknowledgement of the work's authorship and initial publication in this journal.
- Authors are able to enter into separate, additional contractual arrangements for the non-exclusive distribution of the journal's published version of the work (e.g., post it to an institutional repository or publish it in a book), with an acknowledgement of its initial publication in this journal.